|
Veterinaria Tropical. 22(2): 155-169. 1997 DIFERENCIAS GENÓMICAS ENTRE AISLADOS DE CAMPO DE VIRUS Morella de Rolo* e Ivonne Cedeño*
* Investigadores. FONAIAP. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
RESUMEN Ocho ácidos nucleícos (ADN) purificados del virus herpes suis I, recolectados durante 1984-1992 de brotes clínicos de la enfermedad de Aujeszky o Pseudorrabia, en granjas porcinas de Venezuela, fueron comparados mediante análisis de patrones de fragmentos de restricción (PFR), usando la enzima de restricción PVU II y geles de poliacrilamida (PAGE). Los fragmentos del ADN fueron ubicados en regiones especificadas por los rangos de peso molecular (PM) en función al ADN de fago Lambda digerido con Hind III, y ADN de fago O X 174 digerido con Hae III usados como marcadores de PM. La única diferencia detectada entre las cepas de 1984 (C) y 1991 (H) estuvo en un fragmento. El resto de los aislados analizados presentaron diferencias de al menos 3 fragmentos que pueden asociarse con pérdidas o ganancia de sitios de clivaje de la enzima de restricción. Dos aislados colectados del mismo brote con un mes de intervalo presentaron diferencias que pueden asociarse con el pasaje del virus en los cerdos como ha sido señalado previamente por otros autores. Palabras Claves: Enfermedad de Aujesky; enzimas de restricción; variación genómica. INTRODUCCIÓN La pseudorrabia es una infección de suinos distribuida mundialmente, causando grandes pérdidas económicas. En Venezuela se encuentra en todos las zonas productoras de cerdo (ROLO et al., 1993). El agente causal es el virus de la pseudorrabia (PRV) o herpes suis I miembro de los alfa herpesvirinae. Los cerdos son considerados los huesped naturales; sin embargo, una amplia variedad de animales domésticos y salvajes pueden infectarse. La severidad de los signos clínicos y la mortalidad dependen de la edad del huésped y de la virulencia de la cepa. Diferencias en las propiedades biológicas han sido observadas entre aislados de campo (BASKERYILLE y McFERRAN, 1973) y algunos métodos in vitro han permitido diferenciar la virulencia de las cepas (SKODA el al., 1964). El genoma de PRV es una molécula ADN lineal de doble cadena, de aproximadamente 150.000 pares de bases (Pb), lo cual es suficiente para codificar aproximadamente 70 genes. El genoma está dividido en una porción corta (Us) y una porción larga (UI) y dos porciones repetidas IR y TR que se encuentran a ambos lados de la porción corta (METTENLEITER, 1994). Un método para diferenciar cepas virales es usando enzimas de restricción que fragmentan el genoma reconociendo secuencias de nucleótidos específicos para cada enzima, determinando la producción de una serie de fragmentos que se visualizan por electroforesis. Este método ha sido usado para virus como Cytomegatovirus, herpes simple y Epstein Barr . El análisis del ADN aislado de diferentes cepas revela que los patrones electroforéticos de cepas no relacionadas epidemiológicamente pueden variar con respecto al número y tamaño de los fragmentos de restricción de la endonucleasa (GIELKENS y BERNS, 1982, LUDWIG et al., 1982, GIELKENS el al., 1985). En este trabajo se comparó los genomas de diferentes aislados de campo mediante análisis con la enzima de restricción PVU II. Los datos obtenidos indican la existencia de heterogenidad gen ética entre aislados de campo. MATERIALES Y MÉTODOS Cultivo Celular y Virus Las cepas analizadas fueron aisladas de muestras obtenidas en campo durante varios brotes. Las muestras procedían de diferentes entidades federales y años como se observa en el Cuadro 1.
Las muestras de cerebro fueron homogenizadas en medio mínimo esencial (MEM) conteniendo 50 Mg/ml de anfotericina y 8.000 UI/ml de penicilina estreptomicina, manteniendo a temperatura ambiente por 1 hora. La suspensión se inoculó en monocapa de células Vero, fue dejado 1 h a 37 °C para la adsorción, luego se lavó con PBS la monocapa agregando medio de mantenimiento con 3% de suero fetal. Las células se incubaron a 37 °C hasta observar la aparición del efecto citopático (ECP) en el 80% de la monocapa, procediéndose a la cosecha del virus. El virus de referencia usado fue la cepa Bartha, donada por el Departamento de Ciencias Veterinarias de la Universidad de Nebraska-USA, y multiplicada en el laboratorio en monocapa de células Vero. Purificación del Virus El sobrenadante fue recogido al producirse el ECP; se centrifugó a 1.090 g en equipo Beckman modelo J2-21 durante 10 minutos, pasándola a través de un gradiente de sucrosa al 40% en PBS pH 7,2, centrifugando a 10000 g por 4 b a 5 °C; el pellet se suspendió en PBS pH 7,2, manteniéndolo a 4 °C toda la noche. Extracción del ADN Viral Se utilizó una metodología previamente descrita ( PAUL et al., 1982); obteniendo el ADN vital pm extracción con fenol-cloroformo y precipitación con etanol. Los pel1ets se suspendieron en un volumen míinimo de bufer TE(10mm Tris HCl.0,1 mH EDTA, H2O pH 8,0), calculándose la concentración de ADN mediante lectura de densidad óptica a 260 nm (DAVIS et al.,1986) Digestión del ADN can enzima de restricción PVU II y Electroforesis Todas las digestiones se realizaron a 37 °C durante 1 h en el sistema buffer sugerido por la empresa manufacturera. Los fragrnentos obtenidos de la digestión con la enzima PVU II (Promega) fueron sometidos a elecroforesis por el método de geles descontinuos de LAEMMLI (1970) El gel se tiñó por el método de nitrato de plata (DAVIS et al., 1986} Se usaron como marcadores de peso molecular (PM) el ADN de fago Lambda digerido con Hind III y el ADN de fago 0 X 174 digerido con Hae III. Análisis estadístico Para calcular los pares de bases (Pb) de cada fragrnento, se realizó un análisis de regresión aplicando el método de los mínimos cuadrados, a fin de obtener el menor error posible. La recta de regresión se efectuó a partir de las distancias recorridas por los fragmentos, según su peso molecular (P) tanto del ADN de Fago Lambda digerido con la ER Hind III como del ADN Fago 0 X 174, digerido con la ER Hae III, y en los cuales fue calculado y estandarizado el número de Pb en cada fragrnento generado. De esta manera, en todas las corridas electroforéticas se obtuvieron las rectas, patrones que sirven para el cálculo de las distancias de cada banda en términos de Pb Para verificar cuál recta de regresi6n se ajustaba mejor al modelo, se aplicaron ecuaciones: logarítmica, exponencial, lineal y potencial. El ajuste potencial demostró ser el más adecuado, indicando el coeficiente de determinación más próximo a 1. RESULTADOS La enzima de restricción PVU II digirió el ADN de los distintos aislados de virus herpes suis originando entre 40 y 60 fragmentos. Se designaron 4 regiones, sobre la base de los fragmentos producidos por el ADN de Fago lambda digerido con Hind III y el ADN de Fago 0 X 174 digerido con Hae III como patrones. Los límites para cada región quedaron comprendidos así:
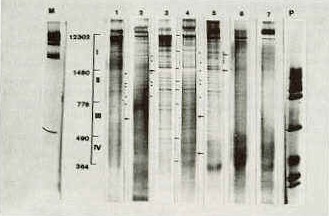
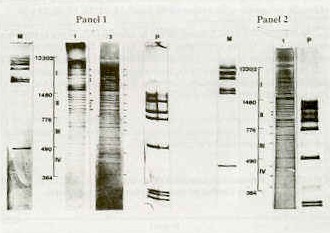
La cepa atenuada Bartha, con deleción en al menos 2 genes digeridos con la misma enzima, sirvió de referencia. Región I Esta región concentra el mayor número de bandas; las muestras C y H presentan 30 fragmentos, permitiendo estandarizar en este número el total de bandas en esta región. Este criterio se utilizó por falta de localización genómica de los fragmentos generados por PVU III en el mapa unidad de PR V. En el gel N° 1 (Figura 1) se compararon las bandas de los aislados con la muestra C y con la muestra G en el gel N° 2 (Figura 2). En cuanto al gel N° 1, la muestra D presenta 27 bandas faltando los fragmentos 18, 19 y 27. Los aislados A y E fueron idénticos con 23 bandas, faltando los segmentos 18, 19,23, 24,25,26 y 27 y con adición de una banda ubicada arriba del fragmento 20. Estas dos muestras son las únicas en presentar esta adición (carril l y 5, Figura 1) .Al comparar B y C, la ausencia de los fragmentos 22, 24, 26 y 27 hace de la muestra B un patrón diferente por ser la única en faltarle el segmento 22.
El análisis de la muestra F fue el más difícil por la poca resolución obtenida, solamente se identificaron las bandas 1,11, 16, 17, 28, 29 y 30. En la Figura 2 gel N°2, la muestra a se diferencia de H por la ausencia del fragmento 11 los segmentos 18, 19, 23 y 24 tienen diferencias de movilidad aunque no significativos estadísticamente. Los fragmentos 20, 21 y 22 presentes en H difieren en G apareciendo 2 pares de fragmentos con patrones de migraci6n diferentes oscilando el primer par entre 2630 y 2670 Pb y el segundo entre 2398 y 2412 Pb. Un resumen esquemático del número de fragmentos se observa en el Cuadro 2. La cepa de referencia Bartha, con 30 fragmentos, se caracterizó por una distribuci6n similar a la muestra H.
Región II En el gel N° 1 (Figura 1) se utilizaron como patrones los aislados C y H para determinar 18 fragmentos en esta región. La muestra A presentó 10 bandas faltando los números 1,3,8,9,11,12,17 y 18. La muestra B con 12 fragmentos no presentó las bandas 3,6,9,12,17 y 18. En la muestra C, con excepción de la banda 1, todas las demás estuvieron presentes quedando 17 fragmentos. En el caso de la muestra D se observan 14 fragmentos faltando los números 1,6,9 y 18. La muestra E presentó ll fragmentos con ausencia de los números 3,8,9, 11, 12, 17 y 18. La muestra F presentó poca resolución por baja concentración de ADN, distinguiéndose 6 fragmentos correspondientes a las bandas 2, 4, 5, 10, 14 y 16. En el gel N° 2 (Figura 2) las muestras G y H difieren en el número de fragmentos; en G se observan 15 bandas con ausencia de 1, 7, 8,9 y U y fue el único en presentar 2 bandas adicionales, una encima del fragmento 6 y otra encima de113. La muestra H con 18 bandas fue la que generó mayor número de fragmentos. La cepa Bartha con 16 careció de las bandas números 8,10 y 17 siendo la única en presentar ausencia de110; además, generó una banda adicional encima del 13 similar a lo ocurrido en G. En el Cuadro 3 se aprecia un esquema del número de fragmentos de las muestras y cepa Bartha.
Región III Los aislados C, E, a y H, así como la cepa Bartha, presentaron 6 fragmentos. Los aislados C y E presentaron migraciones idénticas al igual que G y H. Los valores encontrados en esta región estuvieron comprendidos entre 708 y 512 Pb para C y E, entre 724 y 5l2 Pb para G y H. El aislado D presenta adición de un fragmento de 616 Pb ausente en los demás aislados, conformándose esta región por 9 fragmentos. La cepa Bartha presentó un total de 8 fragmentos con migraciones similares a las muestras de igual número de bandas. En el aislado A se observan 7 bandas desapareciendo el N° 1 y e1 6, lo cual no corresponde al número de pares de bases. Las muestras B y F presentaron baja resolución en esta región. Región IV Las muestras C, E, a y H así como Bartha permanecieron constantes con 6 bandas, migrando entre 467 y 380 Pb para C y E; para a, H y Bartha la migración estuvo entre 446 y 534 Pb. El aislado D fue el único en presentar la adición de 1 banda ubicada entre los dos últimos fragmentos de esta región y calculada en 407 Pb. En el aislado A sólo se observaron 4 fragmentos. Las diferencias observadas entre C y H sólo radican en 1 banda localizada en la región II la cual está presente en H. Estos dos aislados provienen de estados diferentes y presentaron patrones electroforéticos muy parecidos. Las variaciones más frecuentes observadas en la región I fueron los fragmentos 18 y 19 y entre 22-27. La región II fue la que presentó mayor variabilidad y las regiones III y IV fueron las más estables. Las bandas presentes en todos los aislados fueron consideradas regiones constantes del genoma. Las muestras A y E fueron recolectadas durante un mismo brote de pseudorrabia en una granja del Zulia, con un mes de intervalo, la A se obtuvo al inicio y E 30 días después. El análisis indica que sólo en la región I fueron idénticas tanto en número como en movilidad. Las demás regiones presentaron pequeñas diferencias; en E se obtuvieron 4 bandas más que en A por lo cual se consideran aislados diferentes. Las muestras D y G provenían de una misma granja ubicada en Aragua y recolectadas en el mismo momento, ninguna de sus regiones fueron similares, las diferencias encontradas hicieron imposible agrupar estos 2 aislados en un mismo patrón. El aislado D fue el único en presentar adición en las 2 últimas regiones, esto hace pensar en la existencia de 2 cepas diferentes actuando al mismo tiempo. En el aislado B las regiones I y II fueron diferentes al resto de las muestras analizadas por lo cual se ubica como aislado diferente. DISCUSIÓN Se han investigado muchas cepas de PRVen distintos países comprobándose que ciertas secuencias del ADN se encuentran presentes sólo en algunos países; sin embargo, otras cepas tienen una distribución geográfica muy extensa (HERMANN el al., 1984, CHRISTENSEN y SORENSEN, 1988; LOMNICZI, 1991; LUDWIG el al., 1982). A esta conclusión se ha llegado cuando el ADN es digerido con una enzima de restricción que actúa en sitios definidos y conocidos de fragmentación dentro del genoma del PRV. GIELKENS y BERNS (1982) señalaron que utilizando cualquier ER se producen fragmentos constantes producto de la digestión detectados en los patrones electroforéticos. En este caso se observaron una serie de bandas constantes (tomando C y H como referencia) designándolas como partes conservadas entre los aislados. De acuerdo a la distribución de los fragmentos en cada carril, se pudieron observar diferencias marcadas y/o menores al comparar las cepas. Considerando como diferencias marcadas en el ADN de dos cepas, la presencia más de dos lugares de fragmentación, bien por aparición de un lugar nuevo o por desaparición de sitios de fragmentación existente en las muestras patrón (ESPUÑA el al., 1991). En 1982 GIELKENS y BERNS, evaluando 8 aislados digeridos sus ADN con la ER KpnI, observaron que todos fueron diferentes aunque algunos de ellos diferían en 1 sola banda. En nuestro caso, evaluando 8 aislados utilizando EIR PVU II se observó que todos presentaron diferencias en el número de fragmentos obtenidos o patrones electroforéticos, inclusive las cepas C y H que se diferenciaron en 1 sola banda. En 1985 TODD y Mc FERRAN encuentran cepas con variaciones en los patrones de restricción actuando en la misma granja, similar a lo encontrado en este trabajo con las muestras D y G, obtenidas en el mismo momento de inicio del brote. La diferencia entre las cepas A y E, procedentes de la misma granja, pero obtenidas con un mes de intervalo, pudiera deberse al pasaje del virus en los animales de la granja, lo cual coincide con lo expresado por CHRISTENSEN et al. (1987) quienes afirman que los cambios en ciertos fragmentos puede deberse al pasaje del virus en animales. Igualmente WATHEN y PITLE (1984) al realizar análisis electroforéticos de cepas virales después de 11 pasajes seriados en su huésped natural, utilizando los ER Bam HI, SalI, Hinf I y KpnI encontraron variaciones en la migración de los fragmentos probablemente por adición o deleción de secuencias cortas de ADN. En este estudio se demuestra que las cepas A, E y D presentaron bandas adicionales; en el único caso donde pudo haber migración además de adición fue en la cepa G entre los fragmentos 20-22. LUDWING el al., 1982 señalan que cepas no relacionadas epizootiológicamente pueden presentar diferencias y variabilidad de patrones; sin embargo, en este caso las cepas C y H que provienen de entidades geográficas separadas presentaron similitudes muy estrechas. En conclusión, la ER PUV II demostró ser altamente discriminativa para el genoma de PRV, observándose variaciones de cepa de campo del virus PRV en el estudio de 8 genomas. Se confirmó la presencia de 2 variedades genómicas en una misma granja en un mismo brote y al mismo tiempo, se determinó que los perfiles electroforéticos pueden variar debido al pasaje del virus en los animales, al analizar 2 muestras de una misma granja pero tomadas al inicio y luego de un mes de iniciado el brote. SUMMARY Purified DNA from 8 isolates of suid herpesvirus I (SHV-I) collected during 1984-1992 from clinical outbreaks of Aujeszky disease in Venezuelan pig farms, was compared by restriction fragment pattern (RFP) analysis, using PVU II and polyacrylamide gel electrophoresis (PAGE). DNAcleavage fragments were designated in regions specified by molecular weight ranges, based on Lambda phage DNA digest with Hind III, and 0 X 174 phage DNA digest with Hae III as molecular weight markers. The only difference was present in one fragment detected between the strains 1984 and 1991 H. The rest of the analized isolates presented differences in at least three fragments, that might be associated with the virus the passages in pigs as previously reported. Key Words: Aujesky; restriction desease enzimes; genomic variation.
BASKERVILLE, A., J. B. and C. McFERRAN. 1973. Aujeszky's disease in pigs. Veterinary Bulletin 43:465-480 CHRISTENSEN, L. S., K.J. SORENSEN and J. C. LEI. 1987. Restriction fragment pattern (RFP) analysis of genome from Danish isolates of suis herpes virus I (Aujeszky's disease virus) Arch. Virol. 97: 215-224. CHRISTENSEN,L. S. and K.J. SORENSEN.1988.Introduction of foreings strains of suid herpesvirus I (Aujeszky's disease virus) documented by restriction fragment pattern (RFP) analysis. Arch. Virol, 100: 109-113. DAVIS, L. G., M. D. DIBNER and J. F. BATLEY.1986. Basic methods in molecular biology. N. Y. Elsevier Science Publishing 140-400. ESPUÑA, E., F. KOV ACS, I. HEJJA, P. RIERA y B. LOMNICZI. 1991. Variantes genéticas del virus de la enfermedad de Aujeszky en España. Med. Vet. 7 (1): 27-33. GIELKENS, A. L. J. and A. J. M. BERNS.1982. Diferentiation of Aujeszky's disease virus strains by restriction endonuclease analysis of the viral DNA. In: Aujeszky's disease (Current Topics in Veterinary Medicine and animal science). 17:3-13: 1982. Edited by G. Wittmann and S.A. Hall Boston and The Hague: Martinus Nijhoff. GIELKENS, A.L J ., J.T. VAN OIRSCHOT and A. J. M. BERNS. 1985. Genome differences among field isolates and vaccine strains of Pseudorabies virus. J. Gen Virol. 66:69-82. HERMANN, S. C., B. HEPPNER and H. LUDWING. 1984. Pseudorabies viruses from clinical outbreaks and latent infections grouped into four genomes types. In: Latent herpes virus infections in Veterinary Medicine (Current Topics in Veterinary Medicine and Animal Science) 27:37- 401:1984. Edited by Wittmann R., M Gaskell and H. J. Rhiza. Boston and The Haguee: Martinus Nijhoff. LAEMMLI, U. K. 1970. Cleavaje of structural proteins during the assemby of the head of bacteriophage T 4. Nature 227:680-685. LUDWING, H., B. HEPPNER and S. HERMANN. 1982. The genomes o different field isolates of Aujeszky's disease virus. In: Aujeszky's disease (Currents Topics in Veterinary Medicine and Animal Science). 17:15-20:1982. Edited by G. Wittmann and S.A. Hall Boston and The Hague Martinus Nijhoff. LOMNICZI, B. 1991, Estudio molecular de las vacunas de la enfermedad de Aujeszky. Vet. Med. Res. (Hungria) 6-18. METTENLEITER, T. 1994. Pseudorabies (Aujeszky's disease) virus. O.I.E. Symposium Bangkok, Thailand. p 1-9 PAUL, P. S., W. L. MENGELING and E. C. PIRTLE.1982. Differentiation of Pseudorabies (Aujeszky's disease) virus strains by restriction endonuclease analysis. Arch. Virol. 73: 193-198. ROLO, M., L. P ALENCIA, N. LOPEZ , C. MARIN y S. SIFONTES. 1993. Estudio serológico de la Pseudorrabia en Venezuela. Veterinaria Tropical. 18:59-67. SKODA, R., I. BRAUNER, E. SADECKY and V. MAYER. 1964.lmmunization against Aujeszky's Disease with live vaccine. I. Attenuation of virus and some properties of attenuated strains. Acta Virológica 8:1-9. TODD, D. andJ. B. McFERRAN.1985. Restriction endonuclease analysis of Aujeszky's disease (Pseudorabies) virus DNAS comparasión of Northem Irland isolates and isolates from either countries. Arch. Virol 86:167-176. WATHEN, M. W. and E. C. PIRTLE. 1984. Stability ofthe pseudorabies virus genome after in vivo serial passage. J. Gen. Virol. 65:1401-1404. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||